
Contexte du projet
Mise en place le 23 octobre 2000, la Directive Cadre sur l’Eau (DCE) fixe pour objectif l’atteinte du bon état écologique des masses d’eau. Pour évaluer cet état, elle s’appuie sur deux grands volets complémentaires : l’état chimique (concentrations en polluants) et l’état écologique (biologie, hydromorphologie, physico-chimie).
Dans ce cadre, la Fédération de l’Orne pour la Pêche et la Protection du Milieu Aquatique a mis en place dès 2009 un réseau de suivi des cours d’eau fondé sur la méthode des Indices d’Abondance Truite.
En complément du suivi des truitelles, la Fédération de pêche a souhaité, en partenariat avec le Syndicat Mixte de l’Orne et ses Affluents (SYMOA), développer un suivi des populations d’écrevisses à pattes blanches afin d’orienter plus finement les actions de restauration sur les différents cours d’eau du territoire.
En effet, le SYMOA ne disposait pas d’un état actualisé des populations d’écrevisses à pattes blanches avant les travaux. La Fédération de pêche de l’Orne et l’Office Français de la Biodiversité avaient récemment prospecté environ 1 500 stations à l’échelle départementale, mais sur le périmètre du SYMOA, les inventaires dataient de 2001 à 2009. Bien que de nombreuses populations soient supposées disparues, d’autres populations semblaient avoir subsisté. L’étude de diagnostic du Contrat Territorial « Eau et Climat » a notamment permis d’identifier de nouvelles populations potentielles.
En 2025, de nouvelles prospections par suivi ADNe ont ainsi été menées, avec pour objectifs de :
- vérifier la présence ou la disparition des populations historiques,
- confirmer l’existence de nouvelles populations potentielles,
- déterminer des actions de restauration adaptées spécifiquement à cette espèce, lorsque cela s’avère pertinent.
ADN environnemental, kézako ?
L’ADN environnemental (ADNe) correspond à l’ADN extrait directement à partir d’échantillons du milieu (eau, sols, fèces…), sans nécessiter la capture ou l’observation préalable des organismes recherchés. Cette approche permet une détection fine, sensible et non invasive des espèces présentes ou récemment passées dans le milieu.
Zone d’études
À la demande de l’Agence de l’eau Seine-Normandie, il a été décidé, en début d’étude, d’élargir le périmètre d’intervention à l’ensemble du bassin versant de l’Orne, et non plus au seul territoire du SYMOA. Cette décision s’est accompagnée de la mise en place d’un suivi ADNe des mammifères aquatiques, confiée à la Fédération de pêche, afin de répondre à une sollicitation du Groupement Mammalogique Normand.
L’objectif était notamment de cibler la crossope de Miller et la crossope aquatique, deux espèces d’intérêt patrimonial.
© FDAAPPMA 61
© FDAAPPMA 61
Protocoles de l’étude
I) Méthodes d’acquisition des données moléculaires
(Selon Hélène LEBORGNE & Christophe BOUGET, février 2024, Office français de la biodiversité et Muséum National d’Histoire Naturelle)

© SPYGEN
L’ADN contient les informations génétiques propres à chaque espèce. Certaines parties de cet ADN changent très peu d’un individu à l’autre au sein d’une même espèce, mais varient fortement entre espèces différentes. Ces petites séquences particulières, appelées codes-barres ADN, permettent d’identifier clairement une espèce parmi toutes les autres.
Le principe est simple :
1️⃣ On prélève un échantillon
2️⃣ On analyse de très courts fragments d’ADN qui y sont présents
3️⃣ On compare ces fragments à une base de données pour savoir de quelle espèce ils proviennent
Appelée barcoding, cette approche a été utilisée pour analyser l’ADNe de l’écrevisse à pattes blanches, afin de confirmer sa présence ou son absence sur les sites prospectés.

© SPYGEN
Les progrès techniques et la baisse des coûts du séquençage à haut débit ont permis de dépasser les limites du barcoding classique. Au lieu d’identifier l’ADN d’une seule espèce à la fois, les avancées scientifiques permettent désormais de détecter plusieurs espèces simultanément au sein d’un même échantillon.
Cette approche est appelée métabarcoding.
Concrètement, cette technique analyse en parallèle une grande quantité de fragments d’ADN présents dans un échantillon « mélangé » (eau, sol, etc.). Grâce à des outils spécifiques, elle permet d’identifier toutes les espèces dont l’ADN est présent, qu’il s’agisse d’une seule famille d’organismes ou de groupes très variés, selon les marqueurs utilisés.
Il s’agit d’une méthode multi-espèces, très efficace pour dresser un inventaire rapide et précis de la biodiversité locale.
Le laboratoire SPYGEN a utilisé cette méthode pour le suivi des mammifères aquatiques.
Pour couvrir l’ensemble des objectifs fixés, deux protocoles distincts ont été mis en œuvre :
◆ Territoire du SYMOA et de la Communauté de communes des Sources de l’Orne
-
Méthode du laboratoire SPYGEN :– Suivi ADNe des mammifères aquatiques sur 10 stations
– Suivi ADNe de l’écrevisse à pattes blanches sur 4 stations
◆ Ensemble du bassin de l’Orne, du Noireau et de la Rouvre
-
Méthode de la Faculté de Poitiers :– Suivi ADNe de l’écrevisse à pattes blanches sur un total de 50 stations
II) Protocole du laboratoire SPYGEN

1) Échantillonnage de l’eau
Pour garantir des résultats fiables face à la faible concentration en ADNe en milieux courants, SPYGEN a appliqué un protocole précis permettant de maximiser les chances de détection.
À l’aide d’un système de pompage lent et régulier (environ 1 litre par minute) fixé au bout d’une perche, un opérateur a traversé le lit des cours d’eau de berge en berge, en remontant légèrement vers l’amont. La pompe aspire l’eau puis la fait passer à travers un filtre très fin, capable de retenir les fragments d’ADN.
Chaque point d’échantillonnage est filtré deux fois (deux filtres séparés), soit 30 minutes par filtre (ou 30 litres d’eau filtrés).
La crépine, embout qui aspire l’eau, est tenue à distance de toute source de contamination.
Les échantillonnages ont été réalisés lors de conditions hydrologiques stables et hors des périodes de fortes productions végétales (algues, matières en suspension).
2) Analyse des résultats
Les fragments d’ADN ont été amplifiés grâce à la technique de la PCR (une méthode qui « copie » l’ADN pour le rendre détectable).
Chaque échantillon est analysé 12 fois : ce sont les réplicas PCR.
Plus une espèce apparaît souvent dans ces 12 tests, plus sa présence est considérée comme forte ou probable.
1 réplica sur 12 = très faible/trace ponctuelle
11 réplicas sur 12 = forte/régulière
Les résultats sont interprétés en se basant sur deux types d’analyses :
🦞 Analyse VigiDNA S (écrevisses)
Les résultats indiquent simplement si l’espèce est présente ou absente, ainsi que le nombre de réplicas positifs. Cela permet d’évaluer la force du signal.
🐭 Analyse VigiDNA M (mammifères aquatiques)
Les résultats prennent la forme d’une liste d’espèces détectées dans l’échantillon. Pour chacune, on indique le nombre de réplicas positifs, quand cette information est disponible, et le nombre de séquences ADN obtenues, ce qui donne une idée plus précise de l’intensité du signal.
III) Protocole de la faculté de Poitiers

1) Échantillonnage de l’eau
Les prélèvements d’eau ont été réalisés en période d’étiage, lorsque le niveau d’eau était naturellement bas (ici durant l’été 2024). Sur chaque station de suivi, deux échantillons d’eau ont été prélevés à l’aide de bouteilles stériles, en amont et en aval immédiat des radiers, en respectant des protocoles stricts et en portant des gants.
Afin de préserver la qualité des échantillons et l’ADN qu’ils contiennent, l’eau collectée a été immédiatement placée dans une glacière équipée de pains de glace, puis congelée dès le retour du terrain.
Une fois acheminés au laboratoire, les échantillons ont été conservés à -20 °C jusqu’à leur traitement. L’eau a ensuite été filtrée à l’aide d’un système de filtration spécifique, permettant de retenir les fragments d’ADN présents dans le milieu. La filtration s’est poursuivie jusqu’à saturation du filtre, le volume d’eau traité étant systématiquement enregistré.
Les filtres récupérés ont ensuite été conditionnés dans une solution d’éthanol, à l’abri de la lumière et à basse température. Un filtre distinct a été utilisé pour chaque échantillon.
Afin d’éviter toute contamination entre les différents sites de prélèvement, l’ensemble du matériel employé a été soigneusement désinfecté entre chaque station, conformément aux protocoles en vigueur.
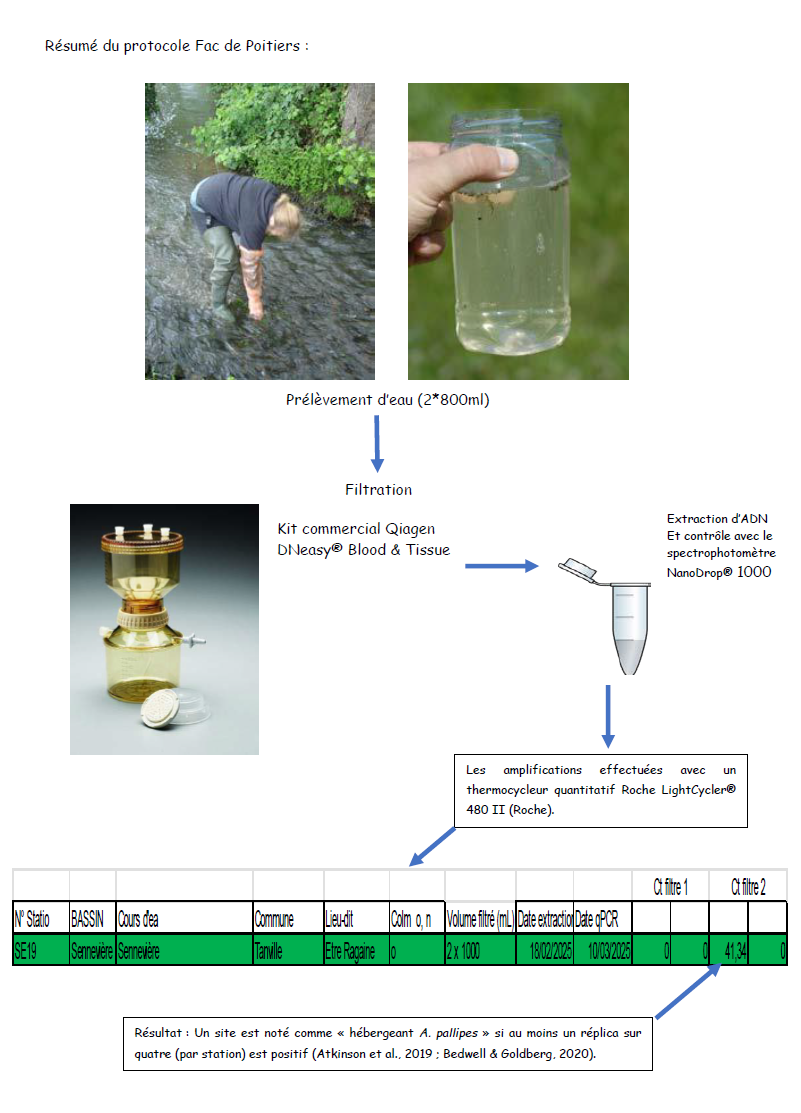
2) Extraction de l’ADN environnemental et protocole qPCR
Les extractions d’ADN ont été réalisées dans une salle dédiée, physiquement séparée des zones de préparation des PCR, afin de limiter les risques de contamination. Le matériel et les surfaces de travail ont été systématiquement décontaminés avant la manipulation des échantillons.
L’ADN a été extrait à partir d’un quart de filtre à l’aide du kit commercial Qiagen DNeasy Blood & Tissue, selon le protocole du fabricant, avec des adaptations décrites par Baudry et al. (2021). L’ADN est élué dans un volume final de 50 µL et conservé à -20 °C jusqu’à son analyse. La qualité et la concentration de l’ADN extrait sont vérifiées par spectrophotométrie.
Pour chaque station, deux échantillons biologiques ont été analysés, chacun en double, soit quatre réplicas par station. Les analyses ont été réalisées par qPCR, dans des espaces dédiés régulièrement décontaminés.
Chaque série d’analyses inclut des contrôles négatifs (eau distillée) afin de détecter d’éventuelles contaminations, ainsi que des contrôles positifs constitués de dilutions d’ADN de référence de Austropotamobius pallipes. Un signal est considéré comme positif lorsque la valeur de Ct est inférieure à 42.
Une station est considérée comme positive pour l’espèce dès lors qu’au moins un des quatre réplicas présente une amplification positive. Cette approche vise exclusivement à déterminer la présence et l’absence de l’espèce, et non à estimer la densité ou l’abondance des populations.
Résultats
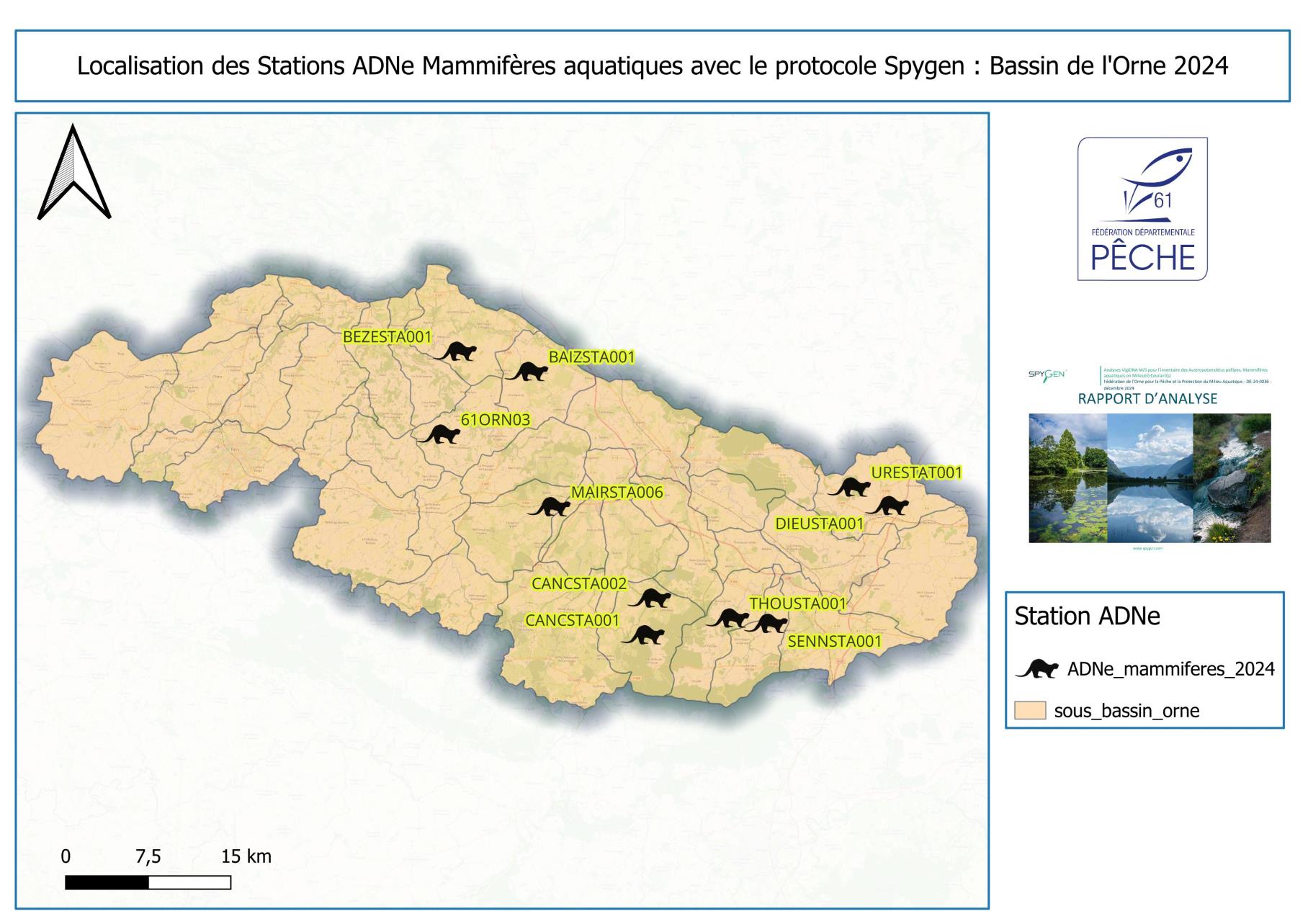
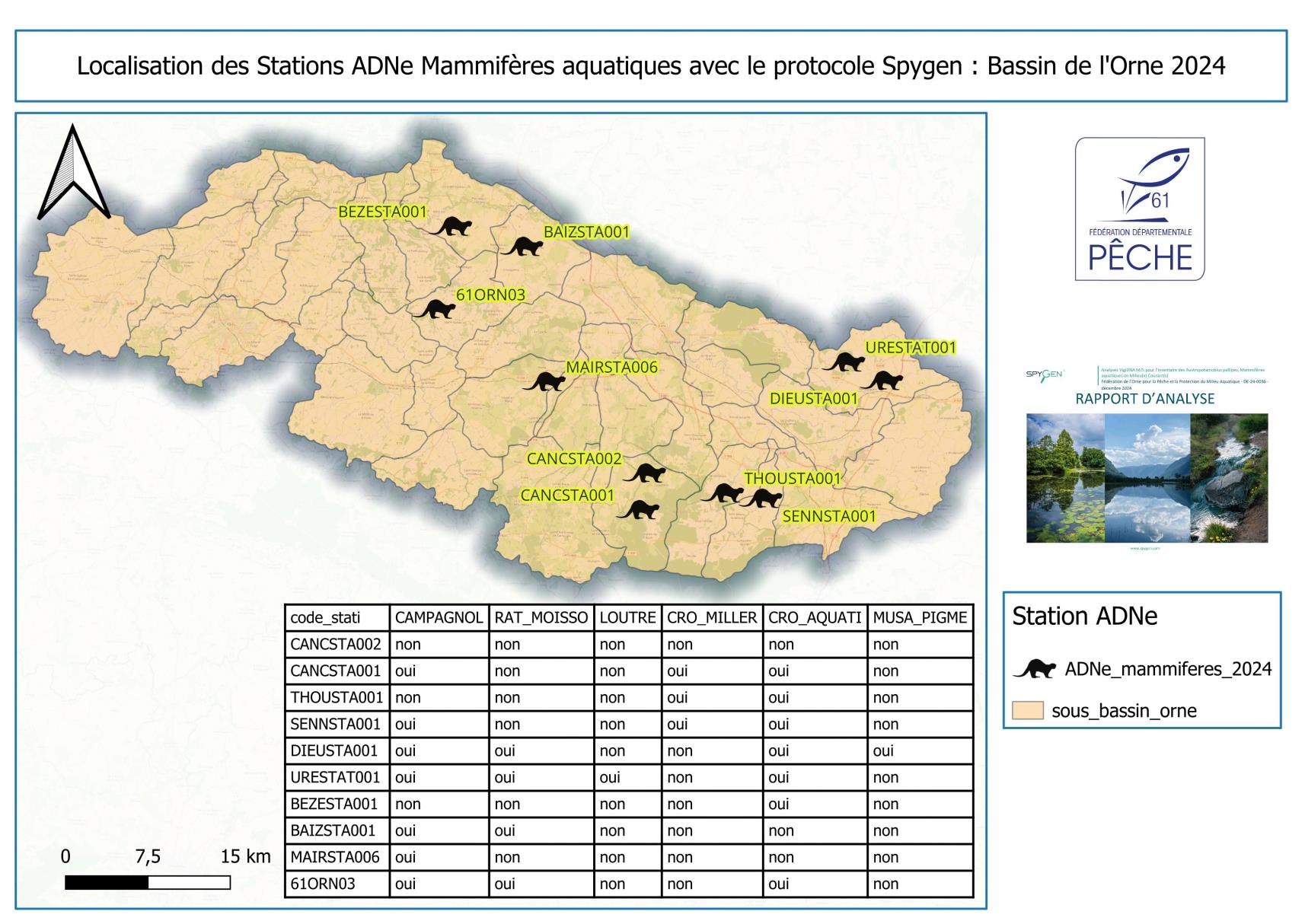
I) Localisation des mammifères semi-aquatiques dans le bassin de l’Orne
Le laboratoire SPYGEN a fourni un inventaire complet des espèces de mammifères détectées sur le site. Cette analyse ne s’est pas limitée aux espèces strictement liées aux milieux aquatiques : elle a également inclus des mammifères terrestres tels que le cheval, le chevreuil, la souris domestique ou encore différentes espèces de chiroptères.
Ces données constituent néanmoins une source d’information précieuse pour le Groupement Mammalogique Normand.
L’étude s’est ici concentrée sur les mammifères semi-aquatiques présentant un enjeu de conservation en Normandie (liste rouge régionale) : le campagnol amphibie, la crossope aquatique, la crossope de Miller et la loutre d’Europe.
© FDAAPPMA 61
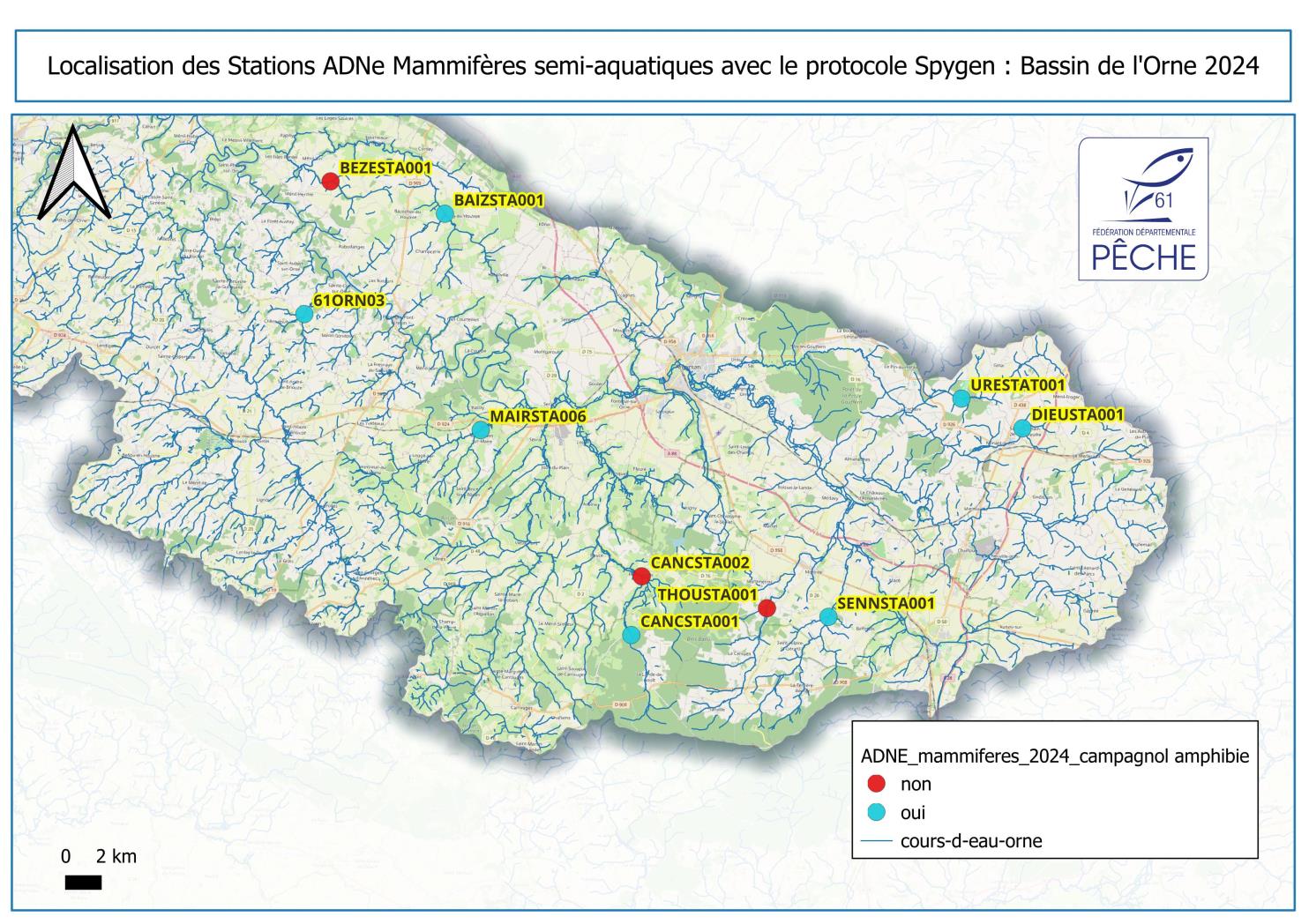

Sur le bassin de l’Orne amont, le campagnol amphibie a été détecté sur sept stations, indiquant une répartition relativement homogène à l’échelle du bassin et une présence vraisemblablement moins rare qu’initialement supposée.
À noter que sur plusieurs stations positives — notamment la Fontaine aux Hérons, la Cance et l’Ure — la ripisylve était relativement dense, ce qui constitue un habitat favorable à l’espèce. Sa présence est donc probable plus en amont du réseau hydrographique.
Toutefois, une prudence dans l’interprétation est nécessaire concernant l’identification spécifique. Dans les résultats fournis par SPYGEN, l’identification des campagnols du genre Arvicola n’est pas systématiquement réalisée à l’espèce. Deux catégories sont distinguées, Arvicola sapidus et Arvicola sp., cette dernière pouvant notamment correspondre à Arvicola amphibius.
À l’heure actuelle, les connaissances régionales indiquent une présence localisée de Arvicola amphibius sur les bassins de l’Yères et de la Bresle, à la limite de la Picardie (Seine-Maritime), tandis que le reste de la Normandie serait majoritairement occupé par Arvicola sapidus.
Cependant, des analyses récentes menées par le Centre permanent d’initiatives pour l’environnement (CPIE) dans les départements du Calvados (14), de la Manche (50) et de l’Orne (61), également réalisées par SPYGEN, mentionnent la présence des deux espèces sur plusieurs stations. Afin de confirmer ou d’infirmer ces résultats, le Groupe Mammalogique Normand prévoit de réaliser prochainement des prélèvements ciblés autour des stations concernées, lesquels seront analysés par un autre laboratoire.
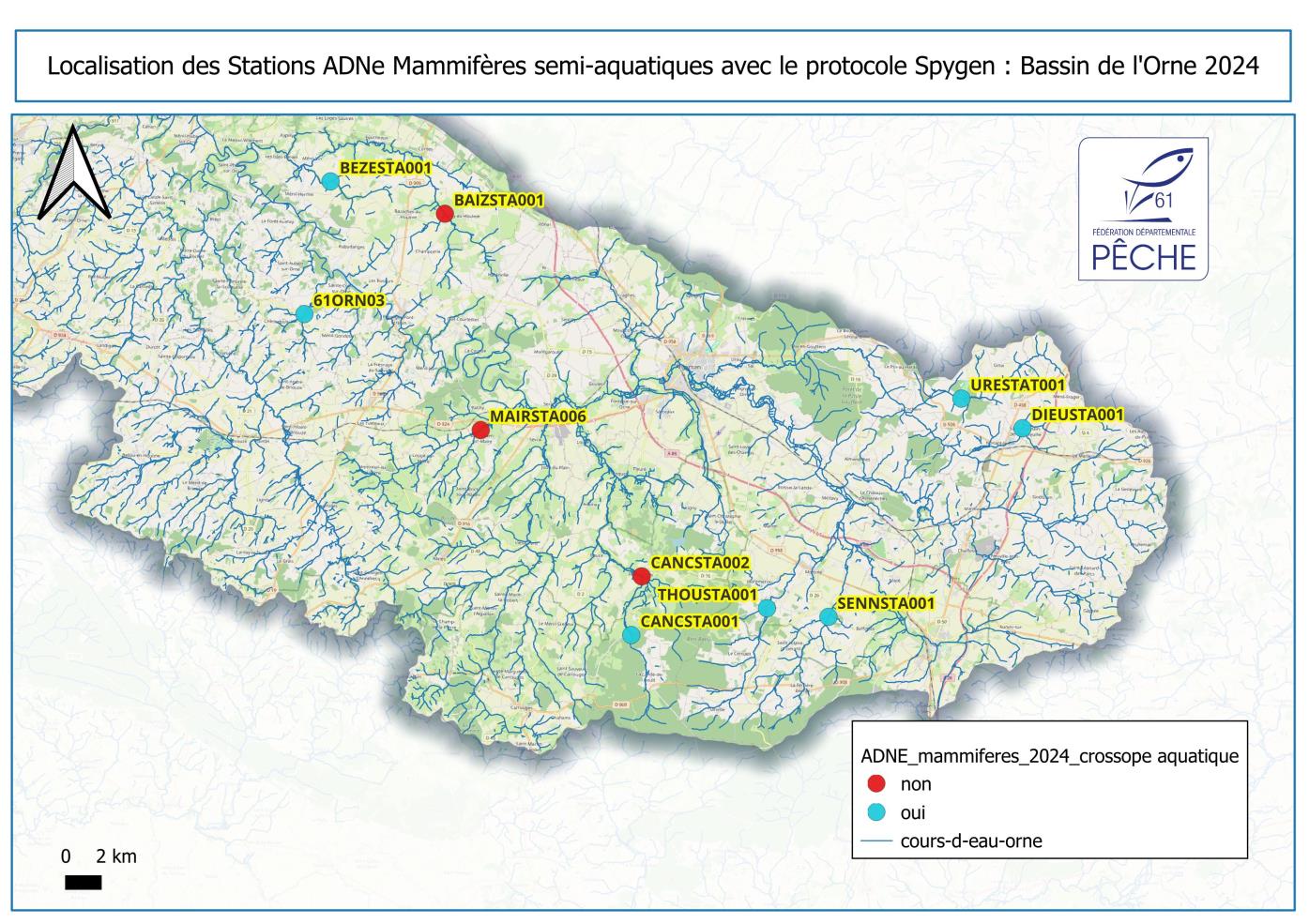

La crossope aquatique a été détectée sur sept stations, témoignant d’une présence bien représentée à l’échelle du bassin.
Elle a notamment été mise en évidence sur le ruisseau de la Fontaine aux Hérons, le Bézeron (affluent de la Baize), la Cance amont, la Thouanne, la Sennevière, ainsi que sur la Dieuge amont et l’Ure.
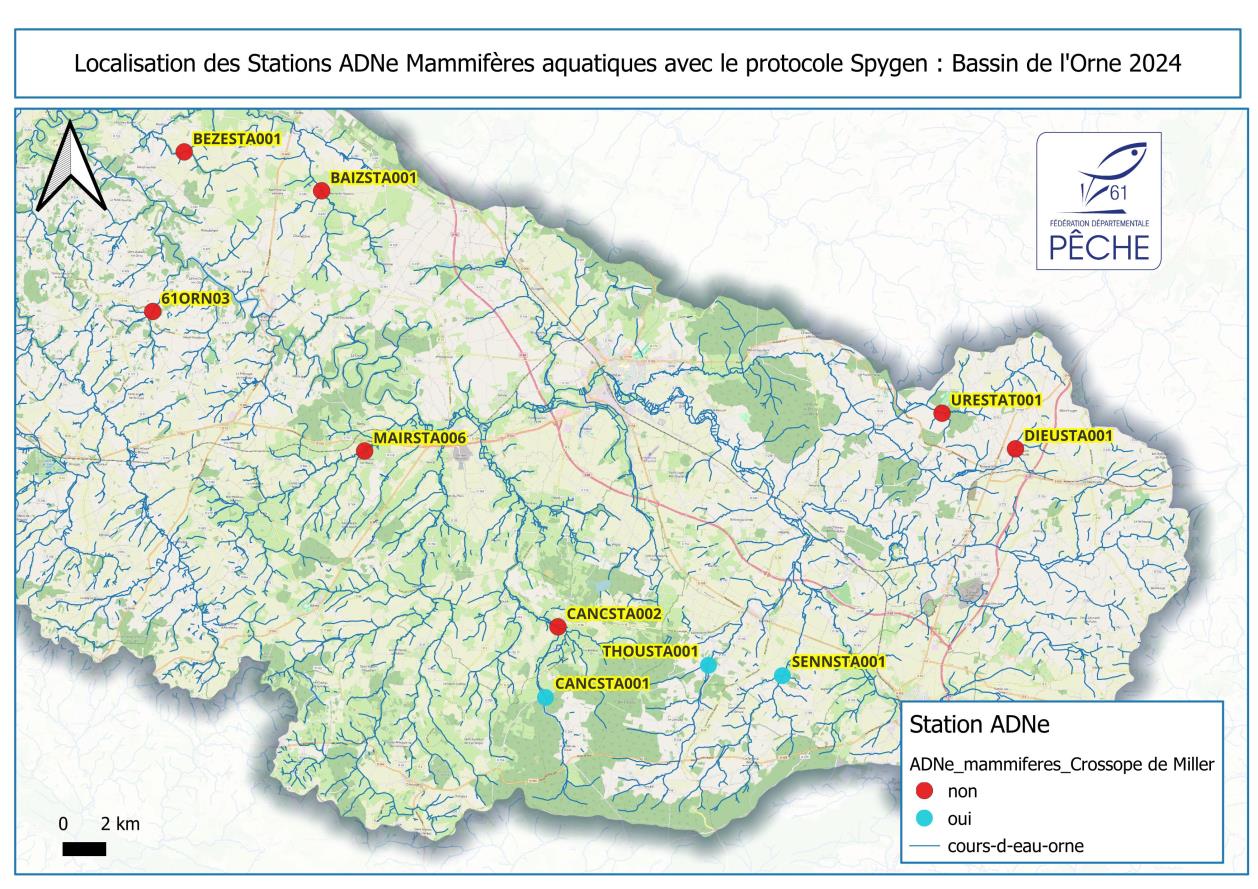

Sur les dix stations inventoriées, seules les trois stations les plus méridionales ont présenté une détection positive de la crossope de Miller.
Ces stations sont situées à des altitudes plus élevées, à proximité du massif forestier d’Écouves, ce qui est cohérent avec la limite de répartition actuellement connue de l’espèce.
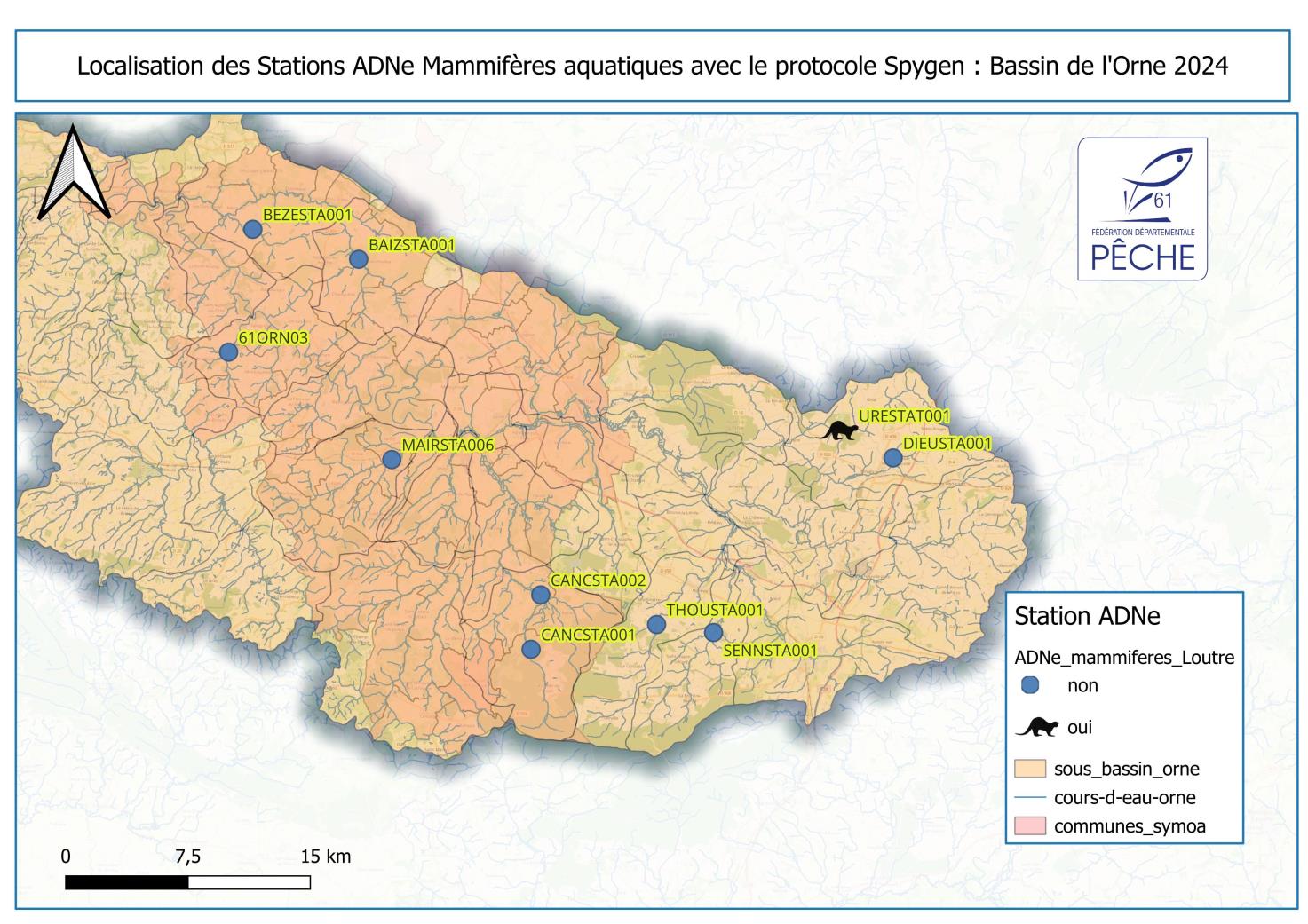

La loutre d’Europe n’a été détectée que sur une seule station : URESTA001.
Cette station étant située sur un territoire appartenant à l’unité expérimentale de l’INRAE, le directeur de l’institut a été informé de cette présence et a relayé l’information auprès des agents en fonction sur le site.
L’absence de détection d’ADNe de la loutre sur les autres stations ne signifie toutefois pas son absence sur le territoire. Le comportement solitaire de l’espèce, associé à l’occupation de domaines vitaux étendus, implique un temps de présence ponctuel dans le milieu aquatique.
Ce mode de vie réduit mécaniquement la quantité d’ADN relâchée dans l’eau et, par conséquent, la probabilité de détection par ADNe.
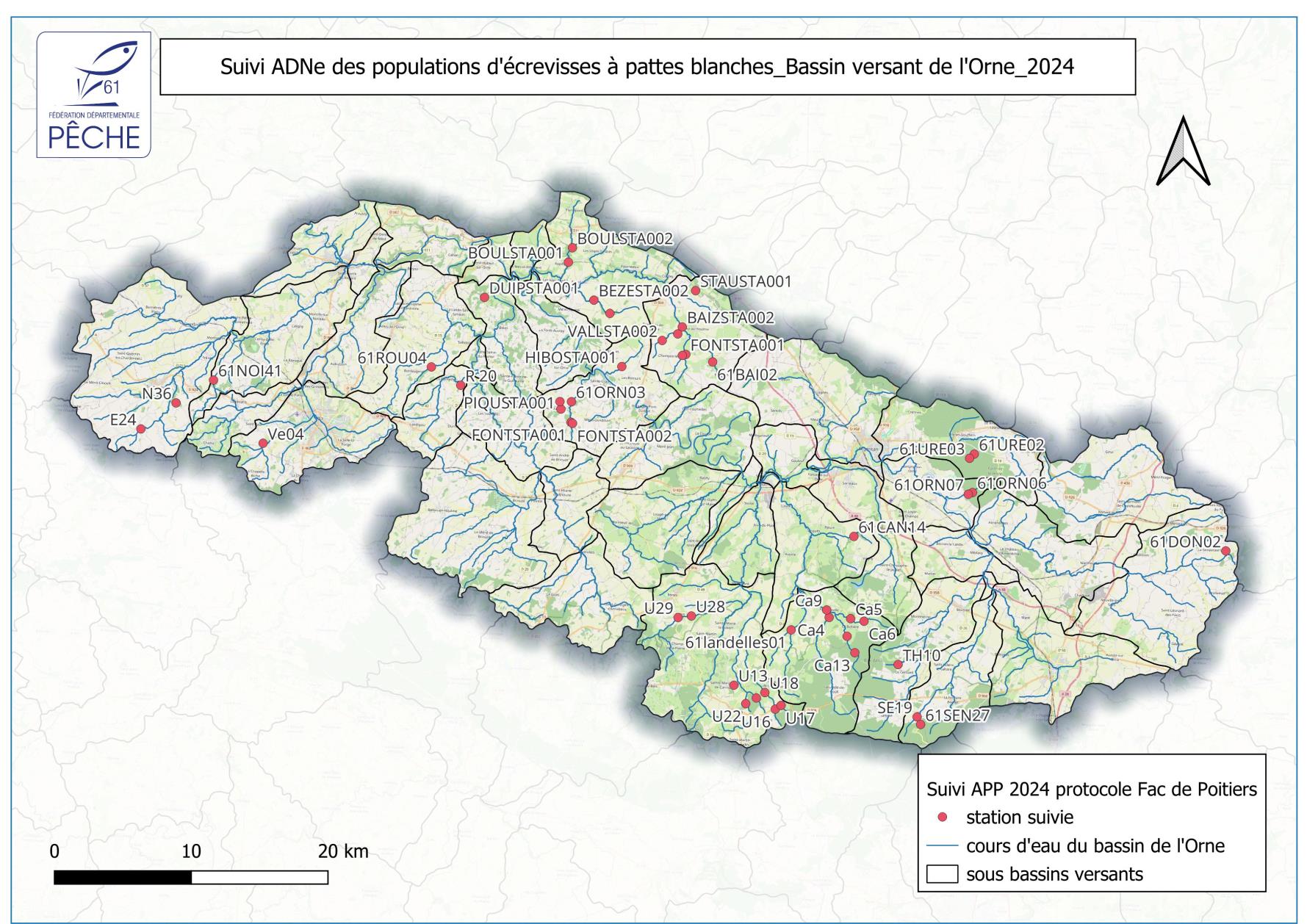
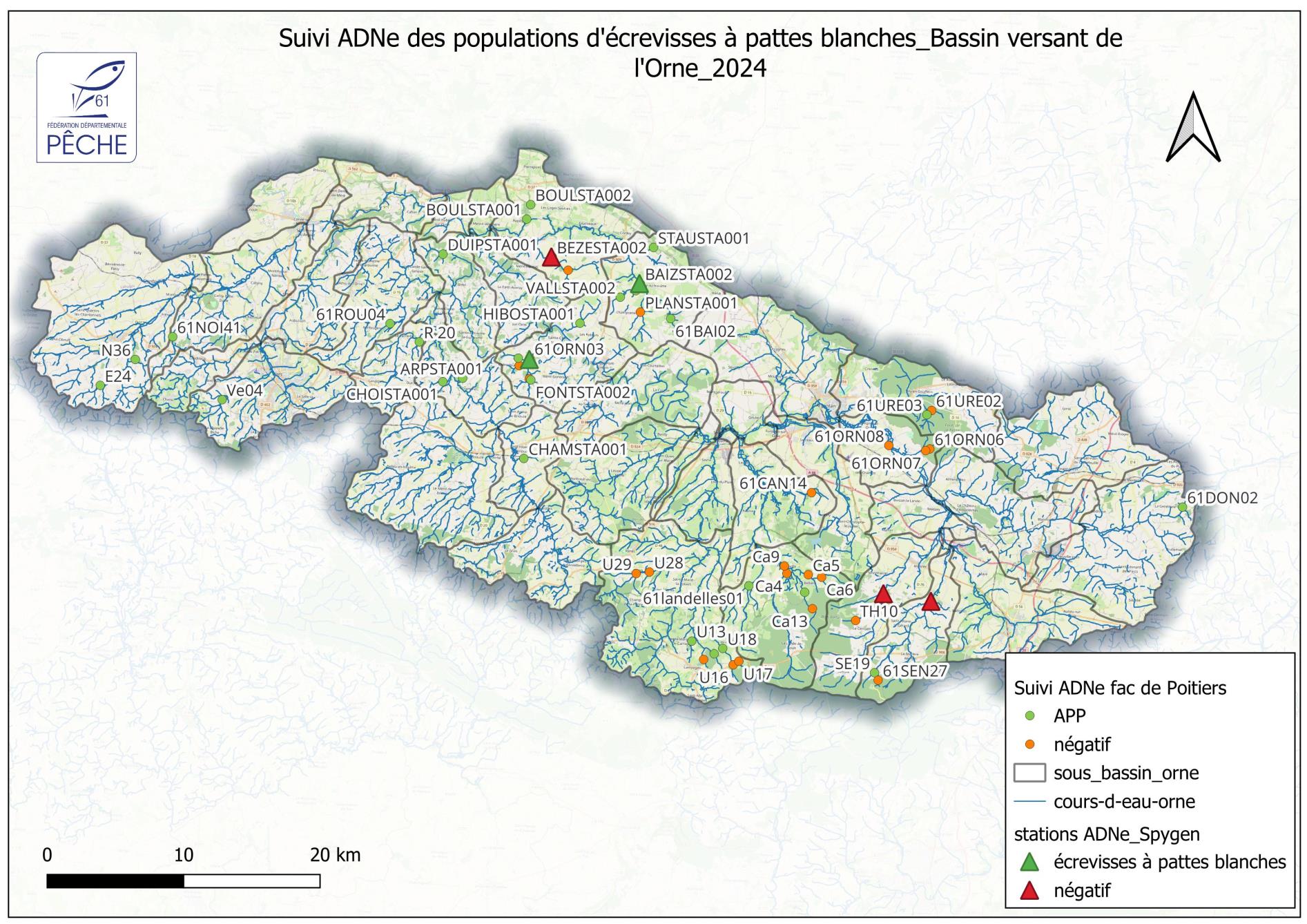
II) Localisation des écrevisses à pattes blanches dans le bassin de l’Orne
© FDAAPPMA 61
L’objectif initial de l’étude était de prospecter exclusivement les stations historiques connues et localisées (50 stations), afin de vérifier la persistance des populations d’écrevisses à pattes blanches et de limiter toute perturbation de l’espèce dans un contexte de travaux. Le Syndicat Mixte de l’Orne et ses Affluents (SYMOA) prévoyait notamment des recharges en granulats et des opérations de reméandrage à proximité de secteurs correspondant à d’anciennes populations.
Il était également pertinent de comparer les deux protocoles de suivi et de compléter les informations de localisation. Ainsi, pour chaque station où le protocole SPYGEN a été mis en œuvre, plusieurs prélèvements complémentaires ont été réalisés plus en amont, selon le protocole de la Faculté de Poitiers.
L’analyse devra être conduite à l’échelle du sous-bassin versant afin d’en tirer des conclusions plus pertinentes et approfondies. Néanmoins, les premiers résultats ont déjà permis de confirmer l’intérêt du protocole d’analyse par ADNe, celui-ci ayant mis en évidence la présence de populations sur des stations où les prospections nocturnes et les inventaires par briques s’étaient parfois révélés négatifs.
Sur les 50 stations prospectées, correspondant à d’anciennes stations à écrevisses à pattes blanches ou de nouvelles stations d’inventaire non référencées, 28 se sont révélées positives, soit 56 % des stations.
Conclusion
Le suivi par ADNe permet un gain de temps considérable et se révèle particulièrement pertinent pour l’identification d’une première localisation spatiale des espèces. Cette approche a notamment permis de mettre en évidence la présence du campagnol amphibie ainsi que des deux espèces de crossopes sur de nombreuses stations. Très discrets, ces mammifères sont difficiles à inventorier par des méthodes classiques, en particulier dans des délais aussi courts.
Le suivi de l’écrevisse à pattes blanches s’est également avéré très efficace, avec 23 populations détectées sur les 36 connues historiquement. La répartition globale de l’espèce apparaît ainsi en diminution à l’échelle du bassin, bien que cette baisse soit moins marquée que celle initialement anticipée. Cette méthode a également permis la découverte de nouvelles colonies, jusqu’alors inconnues.
Le coût attractif et la rapidité de mise en œuvre du protocole développé par la Faculté de Poitiers offrent la possibilité de multiplier les points de suivi et de contrôle, ouvrant la perspective de la mise en place d’une veille départementale « écrevisse à pattes blanches » par ADNe. Une telle démarche suppose néanmoins une excellente connaissance du terrain, une localisation précise des stations et une coordination étroite entre les partenaires.
De son côté, le protocole ADNe mis en œuvre avec le laboratoire SPYGEN apparaît davantage adapté aux suivis multi-taxons. Bien que très fiable, il représente un coût financier et un temps de traitement moins pertinent dans le cadre d’un suivi ciblé sur une seule espèce.
Enfin, la comparaison entre les suivis de terrain (prospection nocturne et méthode des briques) et les analyses par ADNe montre que les trois approches demeurent complémentaires. Dans certains cas, une détection ADNe faible peut être associée à une population pourtant relativement dense. L’ADNe permet ainsi une priorisation spatiale efficace, pouvant être complétée, dans un second temps, par des investigations de terrain plus fines, avec à la clé un gain de temps opérationnel significatif.
Le coût total de l’étude s’élève à 33 737,73 € TTC.
Il comprenait :
- 11 750 € TTC pour le suivi des Indices d’Abondance Truite
- 21 987,73 € TTC pour le suivi ADNe des écrevisses et mammifères semi-aquatiques
Le projet a été financé :
- à 67 %, soit 22 505 € TTC, par l’Agence de l’eau Seine-Normandie
- à 33 %, soit 11 233 € TTC, par la Fédération de l’Orne pour la Pêche et la Protection du Milieu Aquatique et la Fédération Nationale de la Pêche en France






